Lors de la 73e assemblée scientifique et réunion annuelle de la Radiological Society of North America (RSNA) à Chicago, nous avons présenté la première reconstruction en 3D du cerveau d’une personne vivante à partir d’images IRM sous la forme d’un poster et d’une vidéo.
Matériel
Le modèle 3D sous-jacent est basé sur deux ensembles de données d’images provenant de l’imagerie par résonance magnétique (IRM). Chaque ensemble de données se compose de 128 coupes transversales sagittales d’une résolution de 256 x 256 pixels. Ils ont été acquis avec un scanner Siemens Magnetom, en utilisant respectivement les séquences d’IRM Fast Low Angle Shot Imaging (FLASH) et Fast Imaging with Steady-state Precession (FISP).
Méthodes
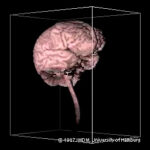 L’environnement de traitement des images a été mis en œuvre sur un mini-ordinateur DEC VAX-11/780 sous VMS avec deux stations de travail d’image connectées (COMTAL Vision One/20, VTE Picturecom). Les contours du cerveau ont été détectés à l’aide d’un opérateur 3D de Marr-Hildreth (chapeau mexicain 3D), ce qui a nécessité environ 230 minutes de temps de calcul, suivies d’environ 45 minutes de correction interactive de certains ponts erronés entre le cerveau et d’autres tissus mous. Les images 3D du cerveau ont été produites avec le programme VOXEL-MAN-8. Le calcul d’une seule image 3D (à droite) à partir du modèle 3D du cerveau a de nouveau nécessité plusieurs minutes de calcul.
L’environnement de traitement des images a été mis en œuvre sur un mini-ordinateur DEC VAX-11/780 sous VMS avec deux stations de travail d’image connectées (COMTAL Vision One/20, VTE Picturecom). Les contours du cerveau ont été détectés à l’aide d’un opérateur 3D de Marr-Hildreth (chapeau mexicain 3D), ce qui a nécessité environ 230 minutes de temps de calcul, suivies d’environ 45 minutes de correction interactive de certains ponts erronés entre le cerveau et d’autres tissus mous. Les images 3D du cerveau ont été produites avec le programme VOXEL-MAN-8. Le calcul d’une seule image 3D (à droite) à partir du modèle 3D du cerveau a de nouveau nécessité plusieurs minutes de calcul.
Résultats
Voici deux courtes séquences de la vidéo numérique originale. Une section un peu plus longue se trouve dans le film Visualiser le corps humain.
Dans la vidéo, l’aplatissement des gyri de l’hémisphère droit causé par une tumeur est clairement visible. Différentes coupes illustrent la localisation de la métastase (en bleu) et l’œdème qui l’entoure. Les valeurs de gris originales des images FLASH et FISP, ainsi que la première composante principale de la transformée de Karhunen-Loeve (KL-TR), sont mappées sur les plans de coupe, soulignant les différents aspects de la lésion.
Ce travail a reçu une mention honorable DAGM de l’Association allemande pour la reconnaissance des formes. Il a servi de base à des modèles et atlas cérébraux 3D ultérieurs, considérablement affinés.
Références
- Michael Bomans, Karl Heinz Höhne, Ulf Tiede, Martin Riemer: 3-D segmentation of MR images of the head for 3-D display. IEEE Transactions on Medical Imaging 9 (2), 1990, 177-183.
- Michael Bomans, Martin Riemer, Ulf Tiede, Karl Heinz Höhne: 3-D Segmentation von Kernspin-Tomogrammen [Segmentation 3D de tomographies par résonance magnétique nucléaire]. In Erwin Paulus (ed.): Mustererkennung 1987, Proc. 9. DAGM-Symposium. Informatik-Fachberichte 149, Springer-Verlag, Berlin, 1987, 231-235.
- Karl Heinz Höhne, Ulf Tiede, Martin Riemer, Michael Bomans, Martin Heller, Gerd Witte: Static and dynamic three-dimensional display of tissue structures from volume scans. Radiology 165 (P), 1987, 420 (Abstract).
Retour à la galerie VOXEL-MAN